サリバチェックラボ 歯周病原細菌検査
測定原理
ポリメラーゼ連鎖反応 (PCR) による増幅を経時的(リアルタイム)に測定することで、増幅率に基づいて鋳型となるDNAの定量を行うことです。PCRとは検出したい菌のある特定の遺伝子配列だけを増幅させる技術のことで、菌種に固有な遺伝子配列を利用した技術です。
口腔内には様々な菌が存在しますが、菌の種類の数だけ遺伝子(DNA)配列が存在します。遺伝子配列はとても長く、この長い領域のなかには違う菌種なのに非常に似通った、もしくは同じ遺伝子配列を持つ部分、また逆に固有な遺伝子配列を持つ部分などがあります。
菌数の測定には、その菌に固有の遺伝子配列部分を認識・結合するプライマー(非常に短い配列)を用います。図1にプライマーを用いたPCRの過程を示します。
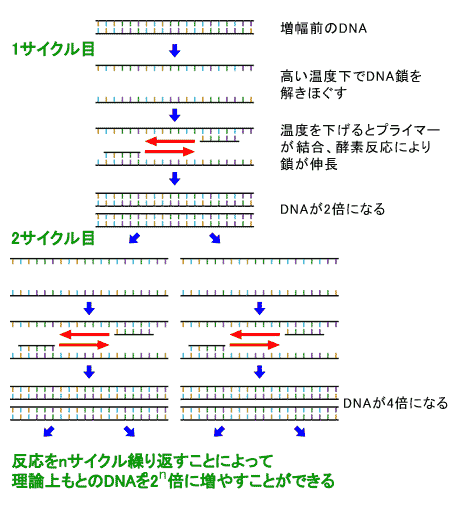
このプライマーは所定の菌にしか結合しないため、口腔内にその菌が存在しないときには、たとえ他の雑菌がたくさん存在しても何も増幅されません。
そして、所定の菌に固有な遺伝子配列が増幅されたことを検出するには、図2に示す技術(TaqMan法といいます)を利用しています。
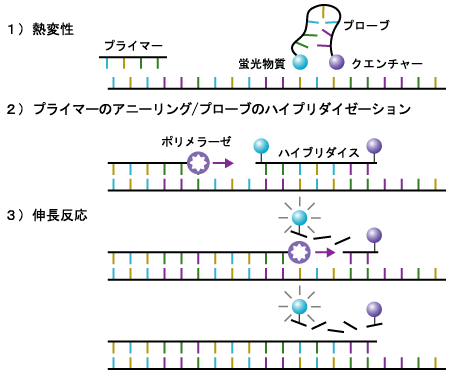
前述のプライマーは、ある領域をはさみこむように設計するのですが、その領域のなかにプローブというこれもまたその菌に固有の配列をもつ非常に短い配列を設計します。プローブには蛍光色素を結合させておき、増幅される過程で蛍光を発する仕組みにしてあります。
図1からわかるように、増幅過程は2倍ずつ増えていくため、指数関数的に増幅産物がつくられます。したがってプローブから発する蛍光量も指数関数的に増加します(図3)。
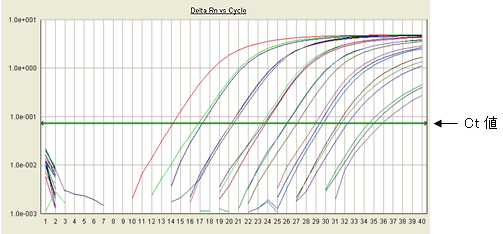 図3
図3増幅はある程度進むとほぼフラットな状態になってしまいます。また、既知のDNA量を増幅させたとき、増幅過程である一定の蛍光量(緑の直線)に達したときの増幅サイクル数(Ct値)とDNA量をプロットすると、直線性を示します(図4)。
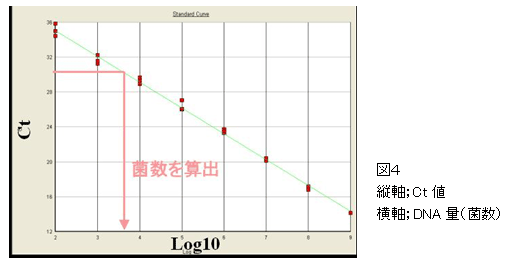
未知のDNA量を増幅させたときのCt値からDNA量が算出でき、定量が可能となります。
P. gingivalis 菌数、A. actinomycetemcomitans 菌数、T. denticola 菌数、T. forsythensis 菌数、
P. intermedia 菌数の定量技術は下記の論文を利用しています。
Yoshida A., Suzuki N., Nakano Y., Oho T., Kawada M., and Koga T. 2003
Development of a 5’ Fluorogenic Nuclease-Based Real-Time PCR Assay for Quantitative Detection of Actinobacillus actinomycetemcomitans and Porphyromonas gingivalis
J. Clin. Microbiol. 41(2): 863-6.
Yoshida A., Kawada M., Suzuki N., Nakano Y., Oho T., Saito T., and Yamashita Y. 2004
TaqMan real-time polymerase chain reaction assay for the correlation of Treponema denticola numbers with the severity of periodontal disease
Oral Microbiology Immunology. 19: 196-200
Suzuki N., Yoshida A., Saito T., Kawada M., and Nakano Y. 2004
Quantitative Microbiological Study of Subgingival Plaque by Real-Time PCR Shows Correlation between Levels of Tannerella forsythensis and Fusobacterium spp.
J. Clin. Microbiol. 42(5): 2255-7
Nagashima S., Yoshida A., Suzuki N., Ansai T., and Takehara T. 2005
Use of the Genomic Subtractive Hybridization Technique To Develop a Real-Time PCR Assay for Quantitative Detection of Prevotella spp. in Oral Biofilm Samples
J. Clin. Microbiol. 43(6): 2948-51